
DataQualityDashboard
DataQualityDashboard は、観察データの品質を評価・可視化するためのオープンソースツールです。
このパッケージは、OMOP CDM インスタンス(現在 v5.4、v5.3、v5.2 をサポート)に対して一連のデータ品質チェックを実行します。
チェックを体系的に実行し、事前に指定されたしきい値と比較評価した後、実行結果を透明かつ分かりやすい方法で報告します。
事前準備
以下のアプリケーションのインストール、設定がされていることが前提です。
「ATLAS セットアップ手順」を実施している場合、下記のアプリケーションはインストール済みとなりますが、時期により推奨されるバージョンがアップデートされている可能性があります。
参考 : https://ohdsi.github.io/Hades/rSetup.html
R環境の設定
以下は、OHDSI R パッケージを実行するために必要な環境をインストールする手順です。
以下の 4つのソフトウェアのインストールが必要です。
1. R
R は統計計算用の環境で、基本的にはコマンドラインのインターフェースを備えたソフトウェアです。
安定性の観点から、今後しばらくは特定のバージョン(2024年6月14日以降は R 4.4.1)をサポート対象としています。
最大限の互換性を得るために、R 4.4.1 のインストールを強く推奨します。
ダウンロード : https://cran.r-project.org/bin/windows/base/old/4.4.1/
2. RTools
RTools は、Windows 上で R パッケージをソースからビルドするために必要なプログラムのセットです。
R と同バージョンをインストールします。
ダウンロード : https://cran.r-project.org/bin/windows/Rtools/rtools44/rtools.html
3. RStudio
RStudio は、R の使用を容易にする IDE(統合開発環境)で、コードエディタ、デバッグ機能、可視化ツールなどを備えています。
快適な R の利用体験のために、RStudio の使用をお勧めします。
ダウンロード : https://posit.co/download/rstudio-desktop/
4. Java(Amazon Corretto 8)
Javaは一部の OHDSI R パッケージのコンポーネント(例:データベースへの接続)を実行するために必要な計算環境です。
ダウンロード : https://docs.aws.amazon.com/corretto/latest/corretto-8-ug/downloads-list.html
さらに、HADES(OHDSI の R パッケージ群)をインストールするには、GitHub の Personal Access Token(個人用アクセストークン)を設定する必要があります。
この手順については、該当セクションで説明します。
ヒープサイズの設定
デフォルトの最大 Java ヒープスペースは非常に小さい (わずか 512MB) です。
Java のヒープメモリ不足エラーを回避するには、最大ヒープサイズを増やす必要があります。
すべての R プログラムに設定するには、.Renviron ファイルに次の行を追加します。
Java では、プログラム実行時に生成されるオブジェクトのためのメモリ領域を「ヒープ領域」と呼び、その領域の大きさを「ヒープサイズ」と呼んでいます。
ヒープサイズを大きくすることで、ガベージコレクションの回数を減らすことが出来るためパフォーマンスの向上に有効ですが、ヒープサイズを大きく設定しすぎるとリソースを無駄に消費したりする原因にもなってしまいます。
R を起動し、以下のコマンドを実行します。
install.packages("usethis")
library(usethis)
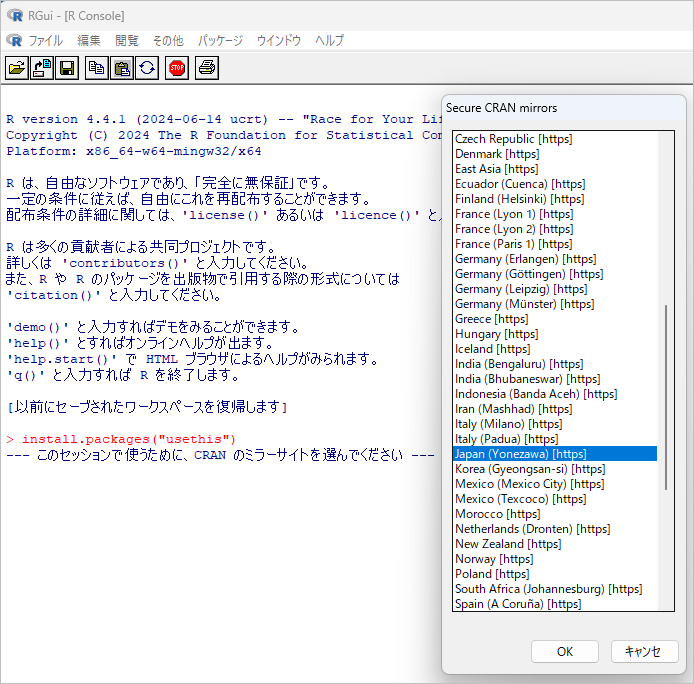
edit_r_environ()ミラーサイトの選択リストが表示された場合、以下が選択候補になります。
- 0-Cloud [https]
- Japan (Yonezawa) [https]
R のエディタ画面が開くので、以下のコードを書き込みます。
_JAVA_OPTIONS='-Xmx4g'
R パッケージをコンパイルするために以下のコードも付け加え、.Renviron ファイルを保存します。
Rtools44_HOME="C:/rtools44"
PATH="${Rtools44_HOME}/usr/bin;${PATH}"※ .Renviron ファイルは、”C:\Users\ユーザ名\Documents” に保存されます
R を再起動します。
これで Java で最大 4GB のヒープスペースが利用可能になります。
インストールの確認
R を起動し、以下のコマンドを実行します。
install.packages("SqlRender")
library(SqlRender)
translate("SELECT TOP 10 * FROM person;", "postgresql")以下の結果が出力されます。
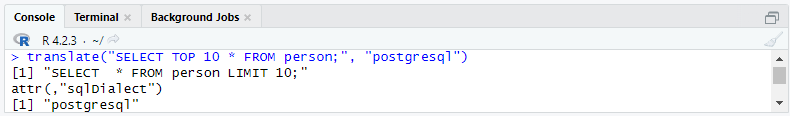
この関数は Java を使用し、R と Java が正しくインストールされていることを確認します。
ソースパッケージのビルドを確認
次のテストは、ソース パッケージのビルドを確認します。
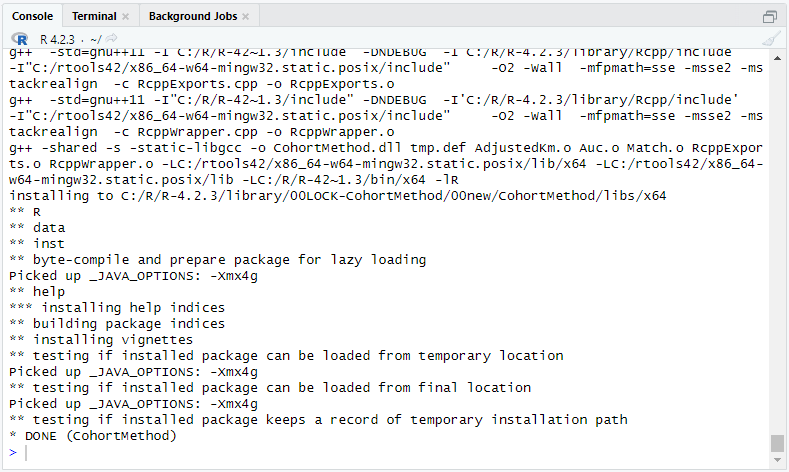
以下の R コードを実行して、OHDSI GitHub リポジトリから CohortMethod パッケージのインストールを確認します。
install.packages("remotes")
remotes::install_github("OHDSI/CohortMethod")以下の画面が表示され、ビルドが確認できました。
GitHub 個人アクセストークン
HADES パッケージをインストールするには、GitHub からのダウンロードが必要です。
しかし、GitHub では匿名ユーザーによる短時間でのダウンロード回数に制限があり、HADES の全パッケージを一括でダウンロードしようとすると、この制限に引っかかってしまいます。
そのため、GitHub アカウントで認証することで、この制限を回避する必要があります。
認証されたユーザーは、多くのダウンロードが許可され、HADES パッケージのインストールが問題なく行えます。
これには、Personal Access Token を作成して R に設定する必要があります。
設定することにより、以後の R セッションでもそのまま使用できます。
PAT(Personal Access Token) の作成手順
まず、有効な GitHub アカウントが必要です。
無料で GitHub.com から作成できます。
usethis パッケージを使ってPATを生成
以下のコードを R で実行すると、PAT を生成するための GitHub のページがブラウザで開きます。
install.packages("usethis")
library(usethis)
create_github_token(scopes = c("(no scope)"), description = "R:GITHUB_PAT", host = "https://github.com")
GitHub へのログインが必要です。
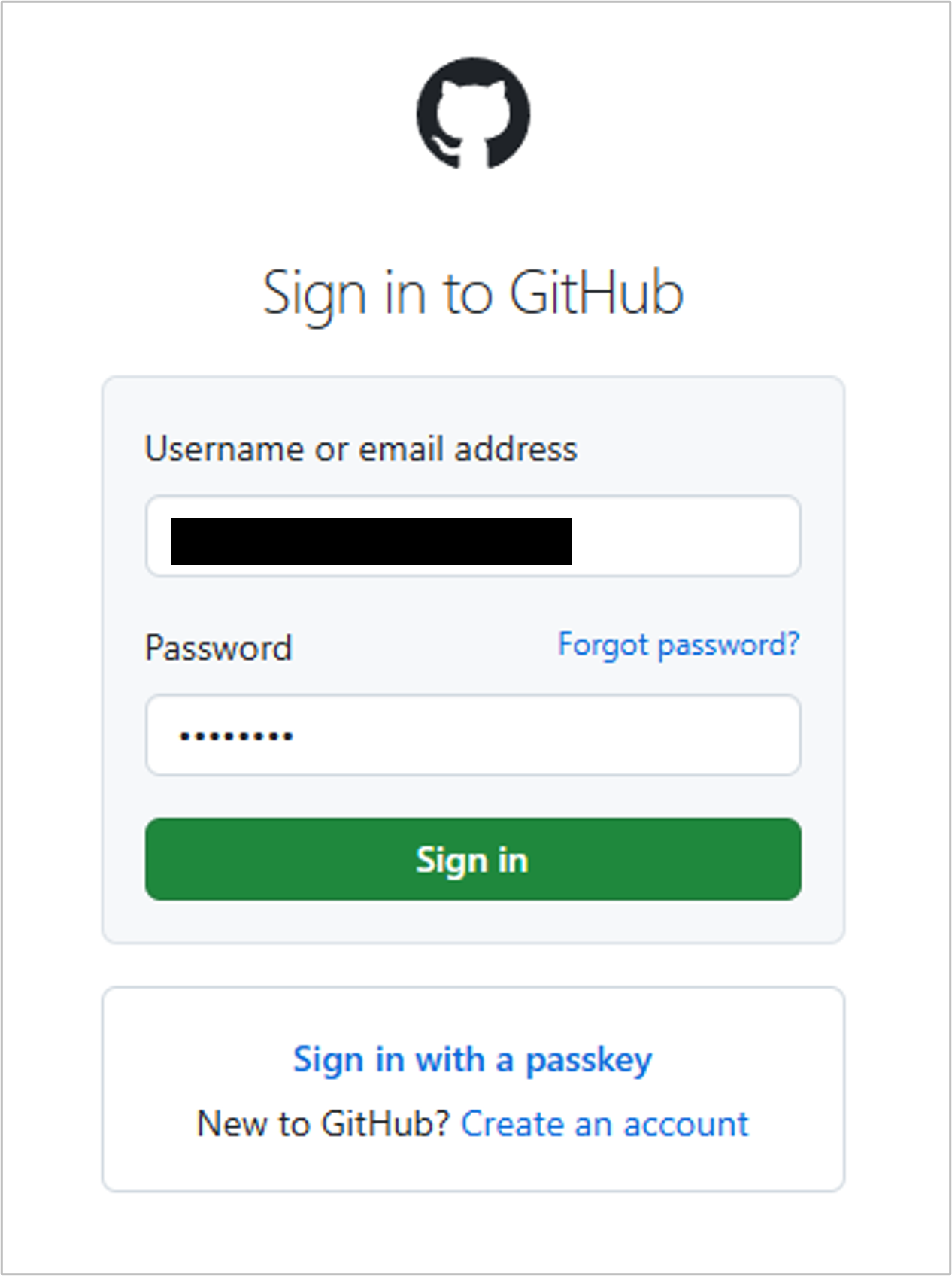
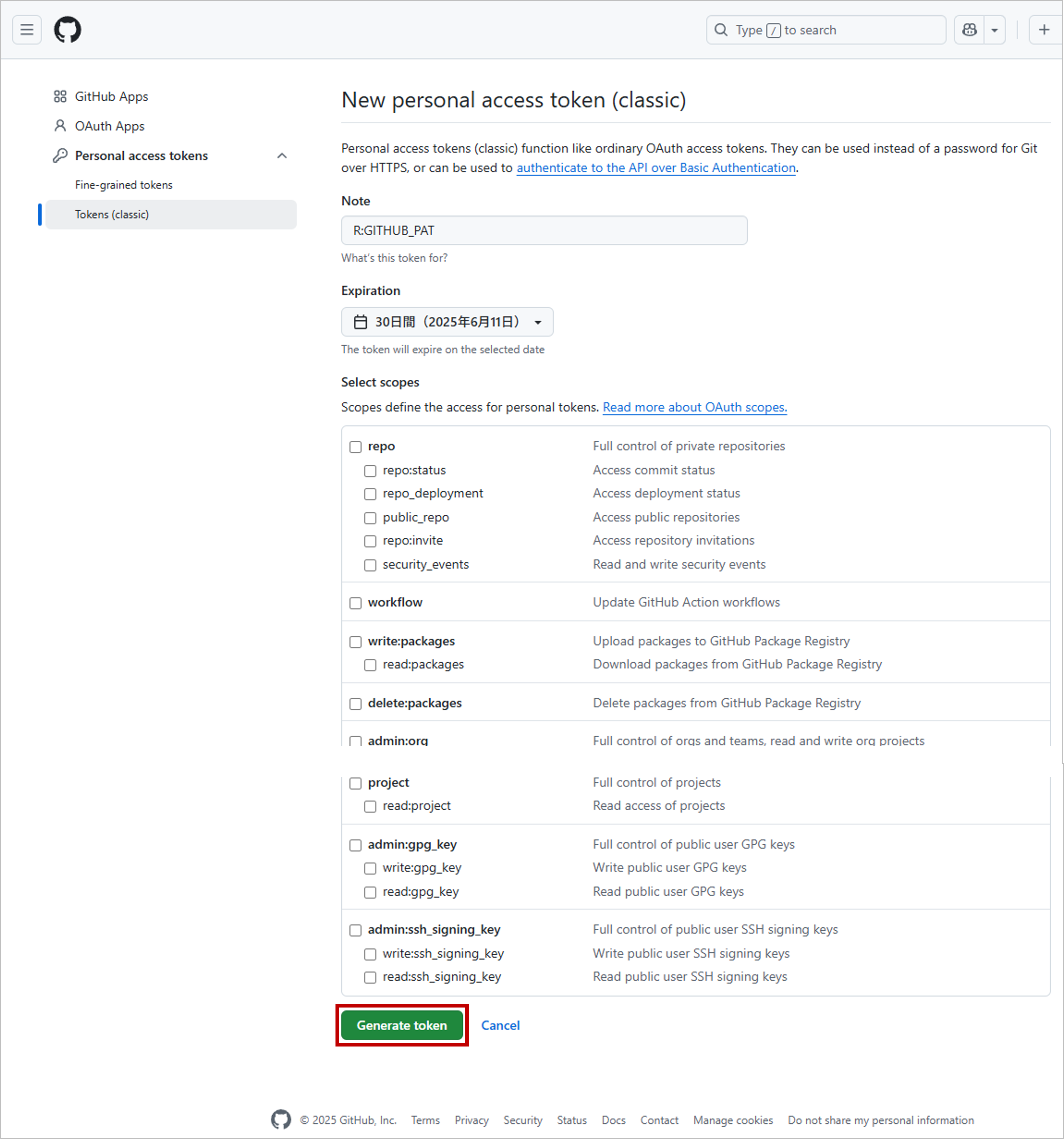
スコープ(アクセス権限)は必要ありませんので、チェックボックスはすべてオフで構いません。
Generate token ボタンをクリックしてトークンを作成し、表示された40文字のトークンを必ずコピーしてください。
※閉じると二度と確認できません
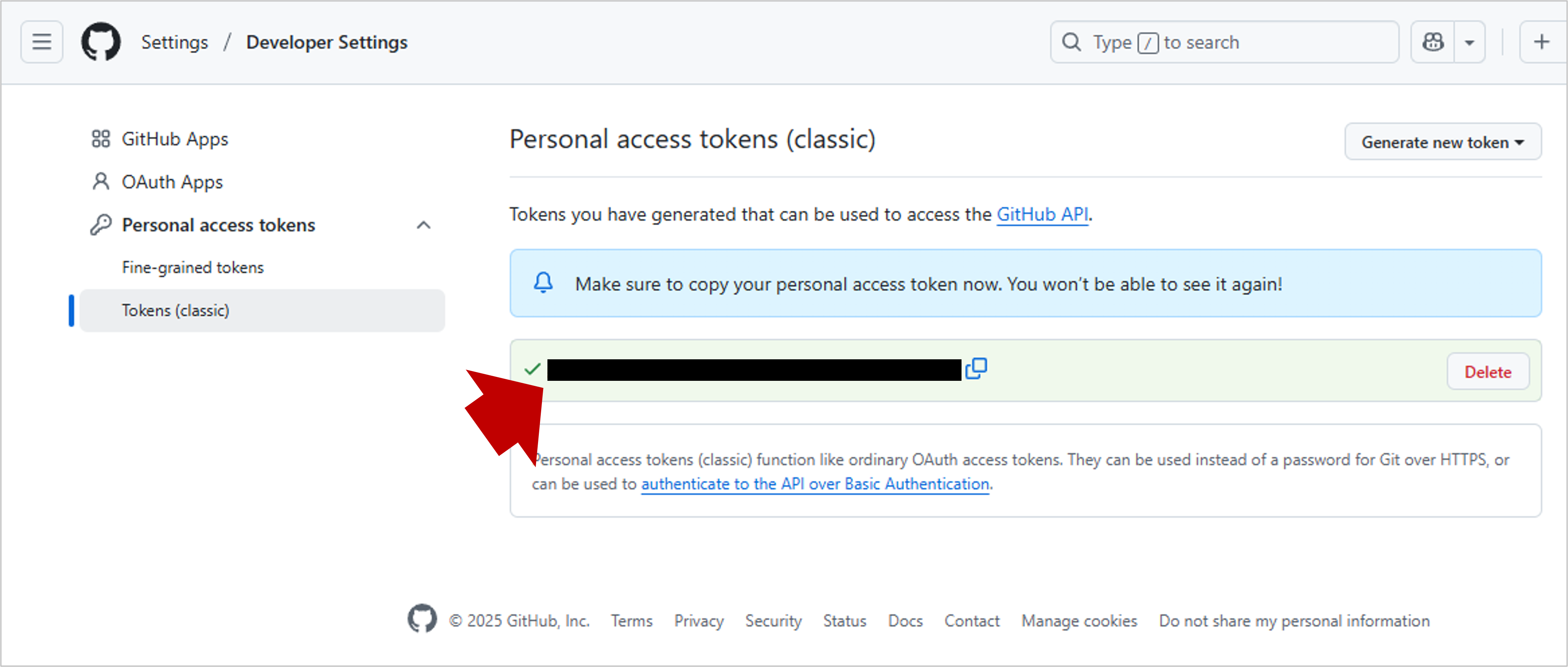
PAT を R に設定する
.Renviron ファイルを開きます。
edit_r_environ().Renviron ファイルに PAT を追記、保存し、R を再起動します。
GITHUB_PAT = 'a1b2c3d4e5f6g7h8g9h0ijklmnopqrstuvwxyz'※'a1b2... の部分は実際の PAT に置き換えてください
これで R 関数から PAT が使用可能になります。
HADES パッケージのインストール
DataQualityDashboard は、HADES の一部です。
HADES の一括インストールを行うことにより、DataQualityDashboard の実行が可能となります。
以下のコードを実行します。
# 「ソースからインストールしますか?」というダイアログを抑制します
options(install.packages.compile.from.source = "never")
# remotesパッケージのインストールと読み込み
install.packages("remotes")
library(remotes)
# HADESのインストール(依存パッケージも含めて常にアップグレード)
install_github("ohdsi/Hades", upgrade = "always")upgrade = "always"を使うことで、依存パッケージをアップグレードするかどうかの確認をスキップできます。
- HADES は依存パッケージの最新リリース版との互換性を前提に作られているため、基本的には常にアップグレードすることを推奨します。
- インストール中に「コンパイルが必要なパッケージをソースからインストールしますか?」と聞かれた場合は、「いいえ(No)」を選択してください。
コンパイルには時間がかかり、また HADES の実行には通常不要です。
DataQualityDashboard の実行
参考:https://ohdsi.github.io/DataQualityDashboard/articles/DataQualityDashboard.html
下記コードをファイルとして保存します。(ファイル名は、DQD.R とします)
※赤文字部分を、実環境のパラメータに書き換えます
# fill out the connection details ----------------------------------------------------------------------- connectionDetails <- DatabaseConnector::createConnectionDetails( dbms = "postgresql", # database version: postgresql user = "ohdsi_app_user", password = "zzzz", server = "localhost/OHDSI", # host/database port = "5432", extraSettings = "", pathToDriver = "C:/Program Files/PostgreSQL" # JDBCドライバーが保存されているフォルダ ) cdmDatabaseSchema <- "your CDM Schema" # CDM schema resultsDatabaseSchema <- "your Results Schema" # Results schema cdmSourceName <- "Your CDM Source" # 任意の名称 cdmVersion <- "5.3" # OMOP CDM version. Currently supports 5.2, 5.3, and 5.4 numThreads <- 1 sqlOnly <- FALSE sqlOnlyIncrementalInsert <- FALSE sqlOnlyUnionCount <- 1 # where should the results and logs go? ---------------------------------------------------------------- outputFolder <- "C:/output" # ファイルの出力先 outputFile <- "results.json" verboseMode <- TRUE writeToTable <- TRUE writeTableName <- "dqdashboard_results" writeToCsv <- FALSE csvFile <- "" checkLevels <- c("TABLE", "FIELD", "CONCEPT") checkNames <- c() tablesToExclude <- c("CONCEPT", "VOCABULARY", "CONCEPT_ANCESTOR", "CONCEPT_RELATIONSHIP", "CONCEPT_CLASS", "CONCEPT_SYNONYM", "RELATIONSHIP", "DOMAIN") # run the job -------------------------------------------------------------------------------------- DataQualityDashboard::executeDqChecks(connectionDetails = connectionDetails, cdmDatabaseSchema = cdmDatabaseSchema, resultsDatabaseSchema = resultsDatabaseSchema, cdmSourceName = cdmSourceName, cdmVersion = cdmVersion, numThreads = numThreads, sqlOnly = sqlOnly, sqlOnlyUnionCount = sqlOnlyUnionCount, sqlOnlyIncrementalInsert = sqlOnlyIncrementalInsert, outputFolder = outputFolder, outputFile = outputFile, verboseMode = verboseMode, writeToTable = writeToTable, writeToCsv = writeToCsv, csvFile = csvFile, checkLevels = checkLevels, tablesToExclude = tablesToExclude, checkNames = checkNames) # inspect logs ---------------------------------------------------------------------------- ParallelLogger::launchLogViewer(logFileName = file.path(outputFolder, sprintf("log_DqDashboard_%s.txt", cdmSourceName))) # DataQualityDashboard -------------------------------------------------------------------- DataQualityDashboard::viewDqDashboard(file.path(outputFolder,outputFile))
実行結果は、output フォルダに保存されます。(保存先は、outputFolder <- "C:/output" を変更することで指定できます)
再度、コマンドを実行する場合は、output フォルダ内のファイルを削除してから実行してください。
DQD.R を RStudio で開きます。
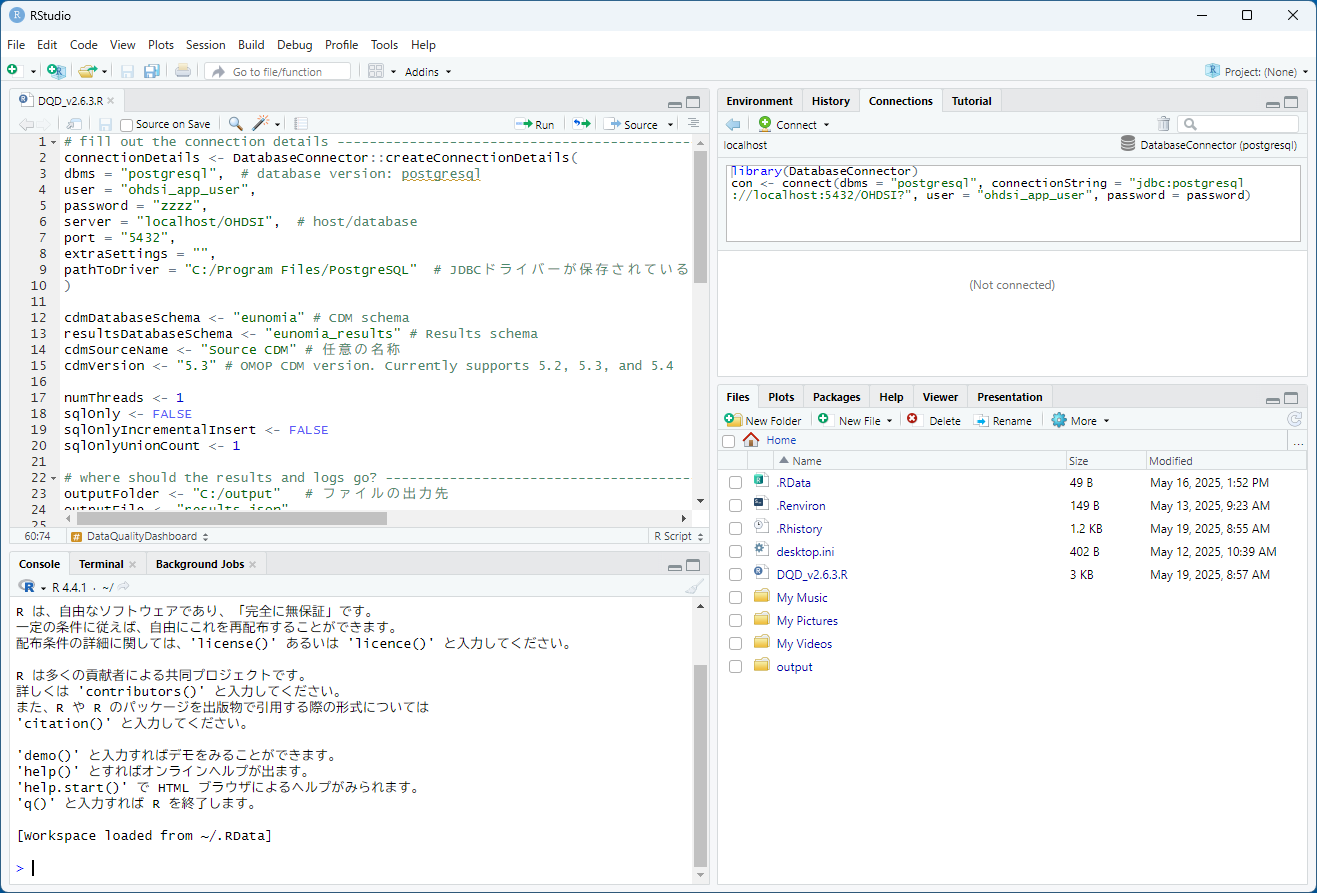
DQD.R の内容が表示され、実行結果が Console 画面(左下)に表示されます。
実行するコマンドを1行目から選択し、”Run” をクリックして実行します。
(動作に問題がなければ、一気に # run the job までのコマンドまで実行しても構いません)
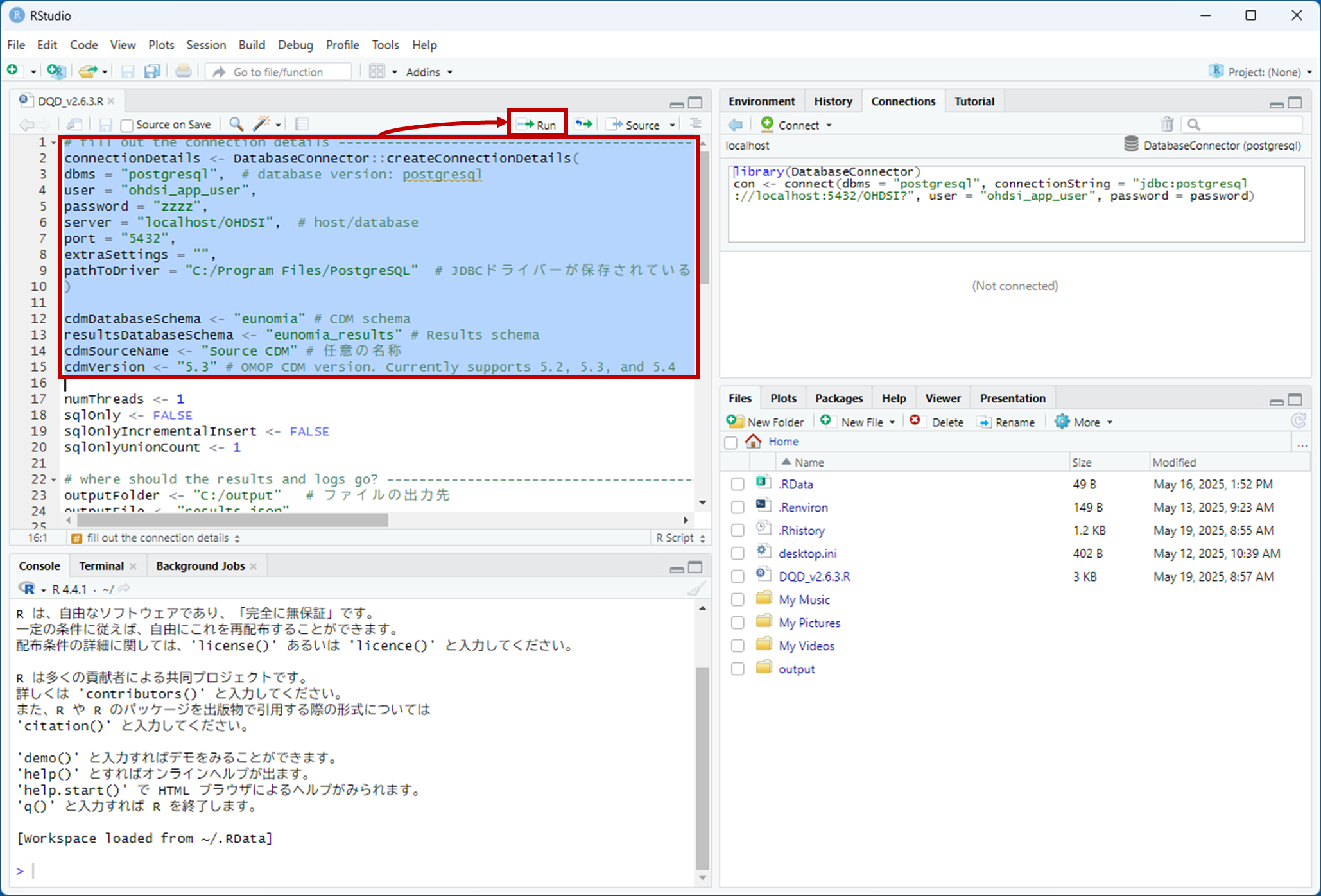
# run the job までの実行が完了すると、DATA QUALITY ASSESSMENT のデータが構築されます。
参考:
DataQualityDashboard のコマンドを実行します。
データ品質の検証結果が閲覧できます。
※下記の画面は、eunomia のデータ品質検証結果です
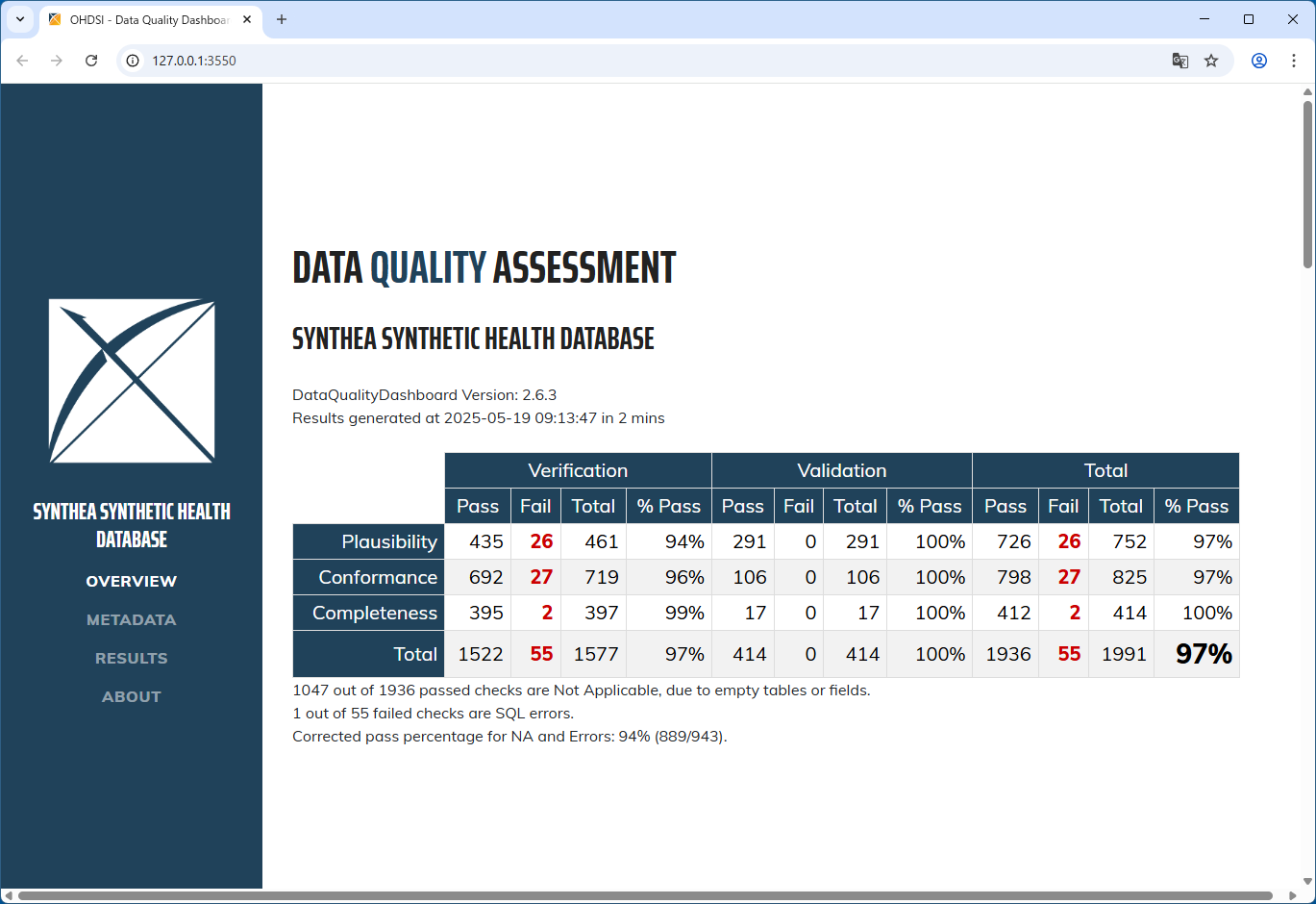
DataQualityDashboard の実行手順は、これで完了になります。
参考情報
DATA QUALITY ASSESSMENT の結果を再度表示したい場合
データ品質の検証結果を再度閲覧する場合、output フォルダにデータが保存されているので、DataQualityDashboard のコマンドを再実行する必要はありません。
以下のコードを実行することにより、保存されているデータ品質の検証結果を表示することができます。
cdmSourceName <- "Source CDM" # 任意の名称
outputFolder <- "C:/output" # ファイルの保存先
outputFile <- "results.json"
# inspect logs ----------------------------------------------------------------------------
ParallelLogger::launchLogViewer(logFileName = file.path(outputFolder,
sprintf("log_DqDashboard_%s.txt", cdmSourceName)))
# DataQualityDashboard --------------------------------------------------------------------
DataQualityDashboard::viewDqDashboard(file.path(outputFolder,outputFile))
データ品質チェック項目
以下の URL にデータ品質チェック項目の詳細が説明されています。
Data Quality Check Type Definitions:
https://ohdsi.github.io/DataQualityDashboard/articles/CheckTypeDescriptions.html
オフライン環境での DataQualityDashboard の実行
オフライン環境では、R パッケージのオンラインリポジトリ(CRAN など)へのアクセスができないため、ライブラリのインストールが直接行えません。
そのため、HADES をインストール済みのオンライン環境からライブラリフォルダを取得し、オフライン環境にコピーすることで、DataQualityDashboard を実行できるようになります。
ライブラリのコピー後は、R にライブラリの検索パスを明示的に指定する必要があります。
以下のように .libPaths() を使用して、ライブラリをコピーしたフォルダのパスを追加して、DataQualityDashboard を実行してください。
# カスタムライブラリパスの追加例
.libPaths(c("D:/library/R-4.4.1",.libPaths()))