テストデータ(Eunomia)のインポート手順
Eunomia は、テストとデモンストレーションを目的とした OMOP CDM の標準データセットです。
Eunomia を OMOP CDM へ適用する手順を記します。
前提条件
OMOP CDM v5.3 のテーブルが構築されていること。
下記 URL の手順を参考に、テストデータ(Eunomia)を取得します。
Eunomia package - RDocumentation
R から以下のコマンドを実行します。
install.packages("Eunomia")
続いて、以下のコマンドを実行します。
library(DatabaseConnector)
library(Eunomia)
connectionDetails <- Eunomia::getEunomiaConnectionDetails()
connection <- connect(connectionDetails)
tables <- getTableNames(connection)
for (table in tables) {
data <- DatabaseConnector::querySql(connection, paste("SELECT * FROM", table))
write.csv(data, paste0(table, ".csv"), row.names = FALSE)
}
disconnect(connection)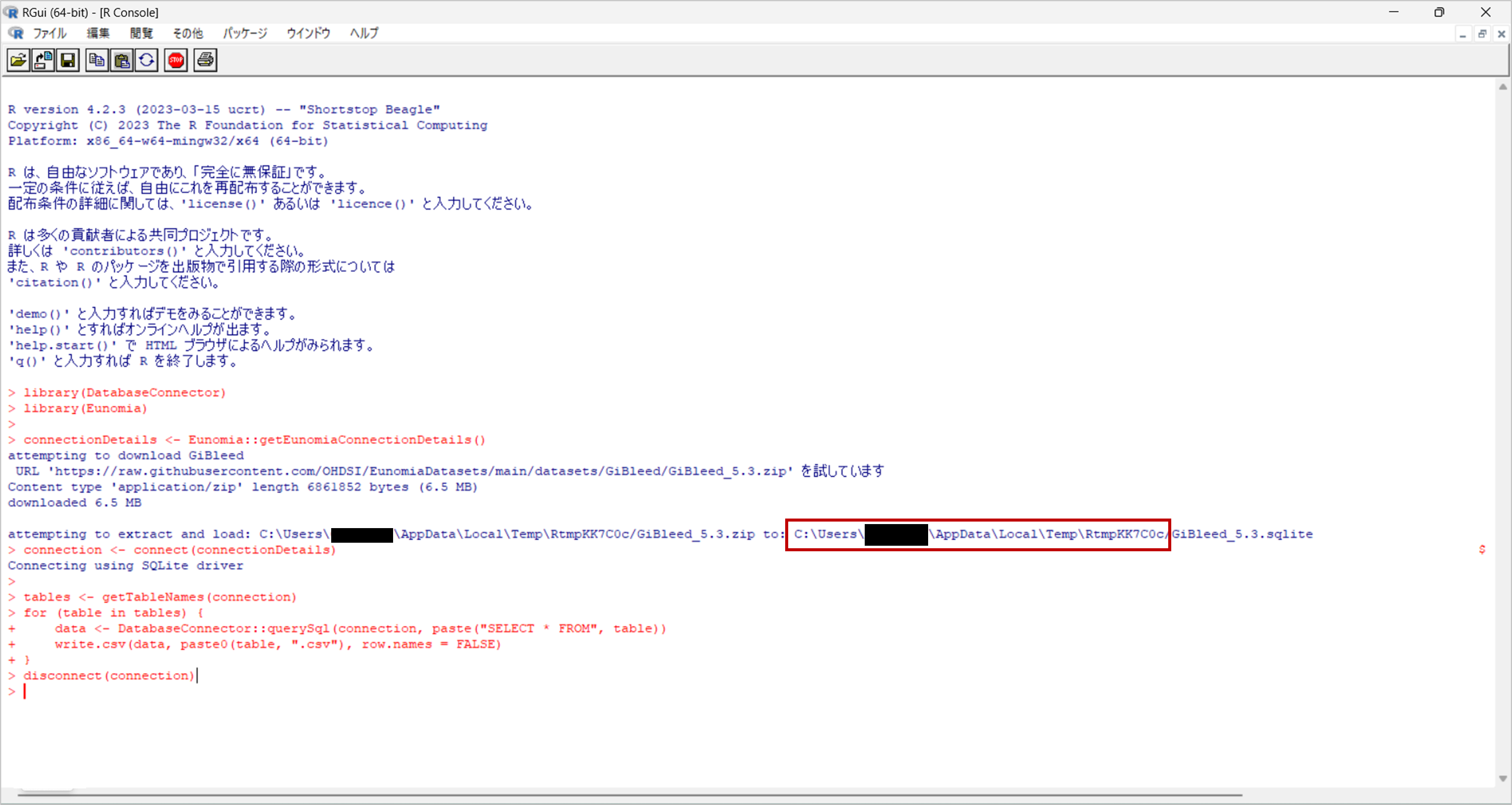
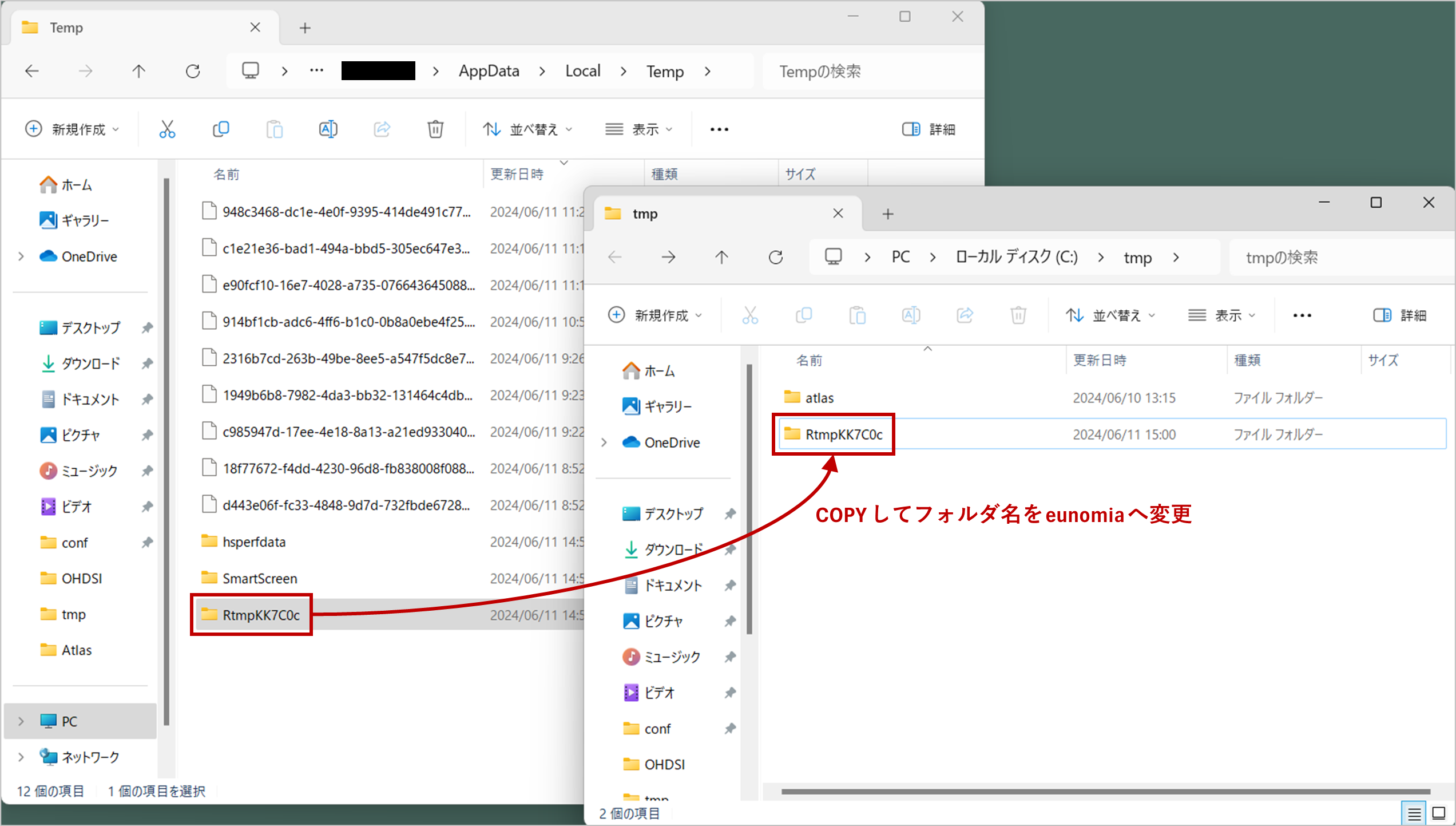
作成された C:\Users\アカウント名\AppData\Local\Temp\RtmpKK7C0c フォルダを C:\tmp へコピーし、フォルダ名を eunomia へ変更します。(RtmpKK7C0c フォルダは、作業毎に違う名称になります)
インポートを始める前に、エクスポートしたデータの内容を一部修正します。
❗
drug_exposure.csv
ファイルをエクセルで開き A 列(DRUG_EXPOSURE_ID)の内容を 1 からの連番に置き換えます。
ファイルをエクセルで開き A 列(DRUG_EXPOSURE_ID)の内容を 1 からの連番に置き換えます。
❗
mesurement.csv
ファイルをエクセルで開き A 列(MEASUREMENT_ID)の内容を 1 からの連番に置き換えます。
ファイルをエクセルで開き A 列(MEASUREMENT_ID)の内容を 1 からの連番に置き換えます。
❗
observation.csv
ファイルをエクセルで開き A 列(OBSERVATION_ID)の内容を 1 からの連番に置き換えます。
ファイルをエクセルで開き A 列(OBSERVATION_ID)の内容を 1 からの連番に置き換えます。
❗
condition_occurrence.csv
condition_occurrence.csv と、OMOP CDM v5.3 の condition_occurrence テーブルの列順が違っています。
インポートする前に、condition_occurrence.csv の列順を修正する必要があります。
最後尾にある “CONDITION_STATUS_CONCEPT_ID” のフィールドを “CONDITION_TYPE_CONCEPT_ID” と “STOP_REASON” の間に移動します。
condition_occurrence.csv と、OMOP CDM v5.3 の condition_occurrence テーブルの列順が違っています。
インポートする前に、condition_occurrence.csv の列順を修正する必要があります。
最後尾にある “CONDITION_STATUS_CONCEPT_ID” のフィールドを “CONDITION_TYPE_CONCEPT_ID” と “STOP_REASON” の間に移動します。
ファイルの修正が完了したら、Eunomia のデータをインポートします。
Git Bash から以下のコマンドを実行します。( zzzz のパスワードは、ohdsi_app_user のパスワードに置き換えてください)
# PostgreSQLの接続情報 export PGPASSWORD='zzzz' PGHOST=localhost PGUSER=ohdsi_app_user PGDATABASE=OHDSI # CSVファイルと対応するテーブル名のリスト declare -A files_tables files_tables=( ["c:/tmp/eunomia/CARE_SITE.csv"]="eunomia.care_site" ["c:/tmp/eunomia/CDM_SOURCE.csv"]="eunomia.cdm_source" ["c:/tmp/eunomia/CONCEPT.csv"]="eunomia.concept" ["c:/tmp/eunomia/CONCEPT_ANCESTOR.csv"]="eunomia.concept_ancestor" ["c:/tmp/eunomia/CONCEPT_CLASS.csv"]="eunomia.concept_class" ["c:/tmp/eunomia/CONCEPT_RELATIONSHIP.csv"]="eunomia.concept_relationship" ["c:/tmp/eunomia/CONCEPT_SYNONYM.csv"]="eunomia.concept_synonym" ["c:/tmp/eunomia/CONDITION_ERA.csv"]="eunomia.condition_era" ["c:/tmp/eunomia/CONDITION_OCCURRENCE.csv"]="eunomia.condition_occurrence" ["c:/tmp/eunomia/COST.csv"]="eunomia.cost" ["c:/tmp/eunomia/DEATH.csv"]="eunomia.death" ["c:/tmp/eunomia/DEVICE_EXPOSURE.csv"]="eunomia.device_exposure" ["c:/tmp/eunomia/DOMAIN.csv"]="eunomia.domain" ["c:/tmp/eunomia/DOSE_ERA.csv"]="eunomia.dose_era" ["c:/tmp/eunomia/DRUG_ERA.csv"]="eunomia.drug_era" ["c:/tmp/eunomia/DRUG_EXPOSURE.csv"]="eunomia.drug_exposure" ["c:/tmp/eunomia/DRUG_STRENGTH.csv"]="eunomia.drug_strength" ["c:/tmp/eunomia/FACT_RELATIONSHIP.csv"]="eunomia.fact_relationship" ["c:/tmp/eunomia/LOCATION.csv"]="eunomia.location" ["c:/tmp/eunomia/MEASUREMENT.csv"]="eunomia.measurement" ["c:/tmp/eunomia/METADATA.csv"]="eunomia.metadata" ["c:/tmp/eunomia/NOTE.csv"]="eunomia.note" ["c:/tmp/eunomia/NOTE_NLP.csv"]="eunomia.note_nlp" ["c:/tmp/eunomia/OBSERVATION.csv"]="eunomia.observation" ["c:/tmp/eunomia/OBSERVATION_PERIOD.csv"]="eunomia.observation_period" ["c:/tmp/eunomia/PAYER_PLAN_PERIOD.csv"]="eunomia.payer_plan_period" ["c:/tmp/eunomia/PERSON.csv"]="eunomia.person" ["c:/tmp/eunomia/PROCEDURE_OCCURRENCE.csv"]="eunomia.procedure_occurrence" ["c:/tmp/eunomia/PROVIDER.csv"]="eunomia.provider" ["c:/tmp/eunomia/RELATIONSHIP.csv"]="eunomia.relationship" ["c:/tmp/eunomia/SOURCE_TO_CONCEPT_MAP.csv"]="eunomia.source_to_concept_map" ["c:/tmp/eunomia/SPECIMEN.csv"]="eunomia.specimen" ["c:/tmp/eunomia/VISIT_DETAIL.csv"]="eunomia.visit_detail" ["c:/tmp/eunomia/VISIT_OCCURRENCE.csv"]="eunomia.visit_occurrence" ["c:/tmp/eunomia/VOCABULARY.csv"]="eunomia.vocabulary" ) for file in "${!files_tables[@]}" do table=${files_tables[$file]} # 外部キー制約を無効化 psql -h $PGHOST -U $PGUSER -d $PGDATABASE -c "ALTER TABLE $table DISABLE TRIGGER ALL;" # 既存データの削除 # psql -h $PGHOST -U $PGUSER -d $PGDATABASE -c "TRUNCATE TABLE $table CASCADE;" # echo "$table テーブルのデータを削除しました。" # CSVファイルのインポート psql -h $PGHOST -U $PGUSER -d $PGDATABASE -c "\COPY $table FROM '$file' CSV HEADER" echo "$file を $table にインポートしました。" # 外部キー制約を有効化 psql -h $PGHOST -U $PGUSER -d $PGDATABASE -c "ALTER TABLE $table ENABLE TRIGGER ALL;" done echo "処理が完了しました。"
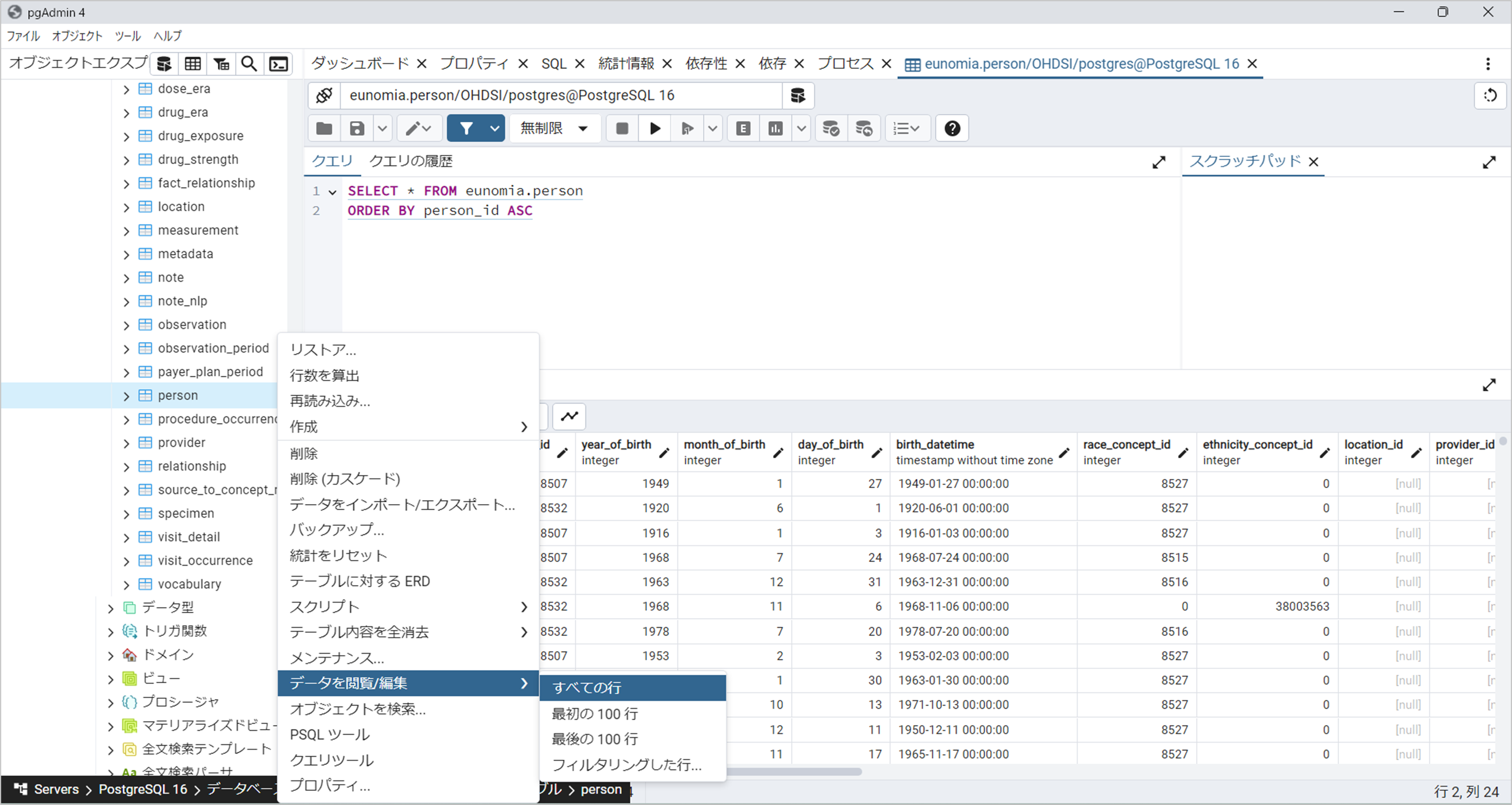
処理が完了し、テーブルにデータが反映されているか確認します。
csv のデータがインポートされていることが確認できれば、テストデータ(Eunomia)のインポートは完了です。